Insbesondere im Zeichen des Klimawandels brauchen wir fundamental neue Konzepte, um umwelt- und klimaverträgliche Energieformen, sogenannte ‘Grüne Energie‘ zu generieren und einzusetzen. Schwerpunkt der AG Photobiotechnologie ist die Erforschung und biotechnologische Anwendung von Wasserstoff-bildenden Enzymen (Hydrogenasen) sowie weiterer Redoxenzyme, welche im mikrobiellen Energiestoffwechsel (wir erforschen vor allem photosynthetische Mikroorganismen) wichtige Funktionen übernehmen oder welche industriell relevante chemische Synthesen katalysieren. Unsere zukunftsorientierte, international vernetzte Forschung mit starkem Bezug zur industriellen Biotechnologie bewegt sich dabei an der Schnittstelle von Mikrobiologie, Biochemie und chemischer Biologie. Zur Aufklärung von Struktur-Funktionsbeziehungen der biotechnologisch relevanten Enzyme benutzen wir eine Vielzahl hochmoderner genetischer, biochemischer und biophysikalischer Methoden und kooperieren national und international mit führenden Wissenschaftler:innen (z.B. Oxford, Osaka etc). Weiterhin erschaffen wir durch Enzym-Engineering auch bioinspirierte neuartige Biokatalysatoren, welche in einer zukünftigen Anwendung im Rahmen der biobasierten Industrie als miniaturisierte, robuste Katalysatoren in H2-Technologien und in der ‘Grünen’ Chemie eingesetzt werden können.
Neben dem Schwerpunkt der Proteinforschung charakterisieren wir Stressantworten von Mikroalgen (siehe auch Hemschemeier). Angesichts des Klimawandels sind in Zukunft häufig extreme Witterungsbedingungen zu erwarten. Wie Pflanzen und Algen sich an diese anpassen, ist von ausschlaggebender Bedeutung für das Ökosystem Erde und unser Klima. Wir erforschen die Anpassung von Mikroalgen an Stressbedingungen, insbesondere Hypoxie, auf physiologischer, genetischer und biochemischer Ebene. Ein Schwerpunkt ist die Erforschung der stress-induzierten Stickstoffmonoxid-basierten Signaltransduktion. Hier charakterisieren wir Häm-bindende Proteine (mikrobielle Hämoglobine und NO-sensitive Guanylatcyclasen) biochemisch und biophysikalisch insbesondere im Hinblick auf NO-relevante Reaktionen. Weiterhin untersuchen wir Signaltransduktionskomponenten und regulierte Zielproteine in physiologischer Hinsicht, indem Interaktionspartner identifiziert und mittels der CRISPR/Cas-Technologie Mutanten der entsprechenden Gene generiert werden. Da einige Mikroalgen als Antwort auf Stress Wasserstoff bilden, ergeben sich hier komplementäre Überlappungen und interdisziplinäre Zusammenarbeit innerhalb der Arbeitsgruppe sowie in unserem internationalen Kooperationsnetzwerk.
Hydrogenasen katalysieren die reversible Reduktion von Protonen zu molekularem Wasserstoff (H2). Sie spielen eine wichtige Rolle im Energiestoffwechsel vieler prokaryotischer und eukaryotischer Mikroorganismen, sind aber auch von großem Interesse für die biobasierte Herstellung und Nutzung von Wasserstoff. Die Evolution hat verschiedene Klassen an Hydrogenasen hervorgebracht, die sich anhand ihrer aktiven Zentren unterscheiden. Wir erforschen die besonders effizienten so genannten [FeFe]-Hydrogenasen, die ein in der Natur einzigartiges aktives Zentrum, das H-Cluster, enthalten: An ein ‚klassisches‘ [4Fe4S]-Cluster ist über einen koordinierenden Cystein-Rest eine Einheit aus zwei Eisen-Ionen gekoppelt, die durch die biologisch ungewöhnlichen Liganden Cyanid und Kohlenstoffmonoxid sowie eine Azadithiolat-Gruppe koordiniert werden (siehe Abbildung). An dieser Einheit findet die eigentliche Katalyse statt. Wir versuchen, die molekularen Mechanismen zu verstehen, die [FeFe]-Hydrogenasen zu solch effizienten Biokatalysatoren machen. Zum Beispiel untersuchen wir mit Hilfe hoch-aufgelösten Kristall-Strukturen und modernen spektroskopischen Techniken wie Infrarot- oder Elektronenspinresonanz-Spektroskopie, wie das Proteingerüst und der anorganische H-Cluster sich gegenseitig beeinflussen. Dabei stellen wir mittels molekularbiologischer Methoden („site directed mutagenesis“) auch Varianten von [FeFe]-Hydrogenasen her, um den Einfluss einzelner Aminosäure-Reste zu untersuchen. So konnten wir beispielsweise dazu beitragen, den konservierten und hoch-effizienten Protonentransport-Weg in dem Proteinmolekül aufzuschlüsseln.
Wir sind auch daran interessiert, wie das H-Cluster in der Zelle assembliert wird. Das [4Fe4S]-Cluster wird nach heutigem Wissensstand durch die Standard-Maschinerie zur Biosynthese von FeS-Clustern hergestellt, während die Herstellung der Zwei-Eisen-Einheit drei besondere Reifungsproteine erfordert, die ausschließlich in [FeFe]-Hydrogenase enthaltenden Organismen zu finden sind. Diese Reifungsfaktoren katalysieren komplexe Reaktionen, um aus Aminosäuren und Eisen das ungewöhnliche aktive Zentrum herzustellen und es anschließend auf das Hydrogenase-Protein zu übertragen.
In lebenden Zellen – aber auch in industriellen Anwendungen – müssen Hydrogenasen mit Redoxpartnern interagieren, welche Elektronen anliefern oder übernehmen. In der Natur sind dies oft kleine Redoxproteine wie Ferredoxine, Cytochrome oder Flavodoxine. Hier sind wir an der molekularen Interaktion der Proteine interessiert und daran, wie in der Zelle Elektronentransportwege koordiniert werden. Besonders interessant sind die [FeFe]-Hydrogenasen in den Chloroplasten einzelliger Grünalgen wie dem Modellorganismus Chlamydomonas reinhardtii. Diese Hydrogenasen interagieren mit dem photosynthetischen Ferredoxin und erhalten unter bestimmten Bedingungen Elektronen aus der photosynthetischen Elektronentransportkette. Hier wird also die licht-getriebene Bereitstellung hochenergetischer Redoxequivalente mit der Produktion von H2 gekoppelt, während in nicht-photosynthetischen Organismen oder industriellen Prozessen energiereiche Substrate oder Strom bereitgestellt werden müssen. Während dieser Stoffwechselweg auch für die Grundlagenforschung und die Ökologie von großem Interesse ist, möchten wir ihn auch für angewandte Prozesse nutzen. Dazu versuchen wir, die H2-bildenden Prozesse in den Algen zu optimieren, aber auch, die Proteine dieser Stoffwechselwege außerhalb der Zelle zu kombinieren und zu optimieren.
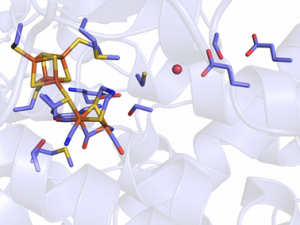
[FeFe]-Hydrogenasen sind äußerst effizient und haben den Vorteil gegenüber Platin-basierten Katalysatoren, dass sie das in großen Mengen auf der Erde vorkommende Eisen nutzen. Bisher ist es jedoch noch nicht gelungen, sie in ökonomisch relevante Prozesse zu integrieren. Ein Grund hierfür ist ihre ausgeprägte Sensitivität gegenüber molekularem Sauerstoff (O2), welcher fast alle bekannten [FeFe]-Hydrogenasen bzw. ihr aktives Zentrum, das H-Cluster, irreversibel zerstört. Ein Schwerpunkt unserer Arbeiten ist es, diese O2-Sensitivität zu verstehen und mittels zufallsgesteuerter („directed evolution“) sowie rationaler Veränderungen am Protein O2-tolerantere Hydrogenasen zu entwickeln. Vor einigen Jahren wurde eine natürlicherweise O2-stabile [FeFe]-Hydrogenasen entdeckt. Wir konnten diese neuartige Hydrogenase strukurell und biochemisch charakterisieren und die Sauerstoffstabilität des Enzyms mechanistisch erklären. Ein Forschungsthema unserer Gruppe ist es nun, weitere O2-stabile [FeFe]-Hydrogenasen zu identifizieren und zu untersuchen, um Enzyme zu entwickeln, die stabil in angewandte Prozesse intergriert werden können.
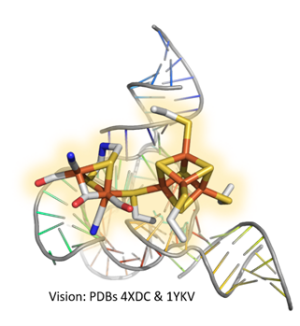
Weiterhin arbeiten wir daran, kleine, robuste Minimal-Hydrogenasen zu entwickeln. Hier kooperieren wir mit Chemiker*innen, die die unter »Molekulare Mechanismen von Hydrogenasen und ihrer Redox-Partner« beschriebene Zwei-Eisen-Einheit des aktiven Zentrum synthetisieren können. In der Tat nutzen wir dies auch, um große Mengen an Hydrogenase-Proteinen in Escherichia coli, dem beliebten Wirt für heterologe Expressionen, herstellen zu können. E. coli besitzt selbst keine [FeFe]-Hydrogenasen und kann daher das aktive Zentrum nicht assemblieren. Wir können aber in dem Bakterium hergestellte Hydrogenasen nachträglich mit der chemisch synthetisierten Zwei-Eisen-Einheit zu aktivem Holo-Protein überführen. Dieses von uns etablierte und überraschend einfache Verfahren erlaubt es uns auch, gemeinsam mit Chemiker*innen veränderte Cofaktoren in das Hydrogenase-Protein einzubauen, um so zu verstehen, welche molekularen Interaktionen einen aktiven Biokatalysator für die H2-Bildung hervorbringen. Mit diesem Wissen versuchen wir nun, das natürliche Proteingerüst stark zu minimieren. Zum einen untersuchen wir, ob das H-Cluster von Minimalpeptiden gebunden und aktiviert werden kann. Eine weitere, möglicherweise futuristisch anmutende Stragetie ist die Nutzung von DNA-Bausteinen zur funktionellen Integration des H-Clusters. Die DNA-Nanotechnologie hat in den letzten Jahren enorme Fortschritte gemacht und kann, neben Mikromaterialien (z.B. DNA-Origami), auch redox-aktive Nukleinsäuren bereitstellen. Hier kooperieren wir mit Wissenschaftler*innen, welche erfahren sind in der Synthese und Manipulation von DNA-Nanostrukturen.
Wir sind davon überzeugt, dass lebende Organismen uns helfen können, sowohl nachhaltige industrielle Prozesse zu etablieren als auch unser Ökosystem zu schützen und zu regenerieren. Insbesondere Organismen, die oxygene Photosynthese betreiben, sind hier besonders vielversprechend. Diese Lebewesen ziehen die zum Leben nötige Energie aus dem Sonnenlicht und benötigen nur anorganische Moleküle – wie CO2, Nitrat oder Sulfat – um die komplexe lebende Materie aufzubauen. Neben unseren Projekten, welche die ‚grüne‘ H2-Bildung durch Algen oder durch isolierte Enzyme aus Algen optimieren sollen, wollen wir weitere komplexen Biosynthesewege aus photosynthetischen Mikroorganismen für biobasierte Syntheseprozesse nutzen. Ein Beispiel sind so genannte Old Yellow Enzymes. Diese flavin-haltigen und dadurch gelb gefärbten Enzyme sind En-Reduktasen, die eine Vielzahl von Redoxreaktionen katalysieren. Außerhalb der lebenden Zelle können sie viele Moleküle herstellen, für die es bereits einen industriellen Markt gibt, wie etwa Duftstoffe oder Insekten-abwehrende Substanzen. Diese Enzyme gibt es ebenfalls in Algen und wir charakterisieren die Proteine, um möglicherwiese neue oder optimierte Eigenschaften zu entdecken. Auch diese Enzyme benötigen eine Elektronenquelle, und zwar das bekannte Redoxäquivalent NADPH. Möglicherweise von größerem Interesse ist es daher, dass wir entdeckt haben, dass auch lebende Algenzellen die entsprechenden Stoffe umsetzen können, und dass diese Biotransformation von der Photosynthese abhängt. Daher erforschen wir die Funktion und metabolische Integration der Old Yellow Enzymes in Algen. Zusätzlich zu der klaren Perspektive für eine biotechnologische Anwendung interessieren wir uns für die physiologische Funktion dieser Enzyme in Algen, denn darüber ist bisher so gut wie nichts bekannt.
siehe Forschungsschwerpunkt Hemschemeier
